optunaでlightgbmのハイパラメータチューニングを実施
今回はoptunaを用いてlightgbmのハイパラメータチューニングを行いました。データはsklearnの乳がんデータセットを用いました。
※このブログの内容は個人の意見・見解となります。また、記事の内容の正確性については保証いたしません。本ブログの目的は分析結果をアウトプットしていくことによる自身のデータ分析力向上を主としています。誤りや違うアプローチの方が良いという事も多分にあると思います。後学のため、「もっとこうしたらいい」や誤りを見つけた場合はコメント欄などでお知らせいただけると有難いです。
○optunaについて
optunaには自動ハイパラメータチューニングフレームワークになります。最適なハイパラメータの組み合わせを見つけるためにベイズ最適化アルゴリズムを使用しています。githubにlightgbmやcatboost、keras、pytorchなどで簡単に使用できます。
GitHub-optuna : https://github.com/optuna/optuna
○データの準備
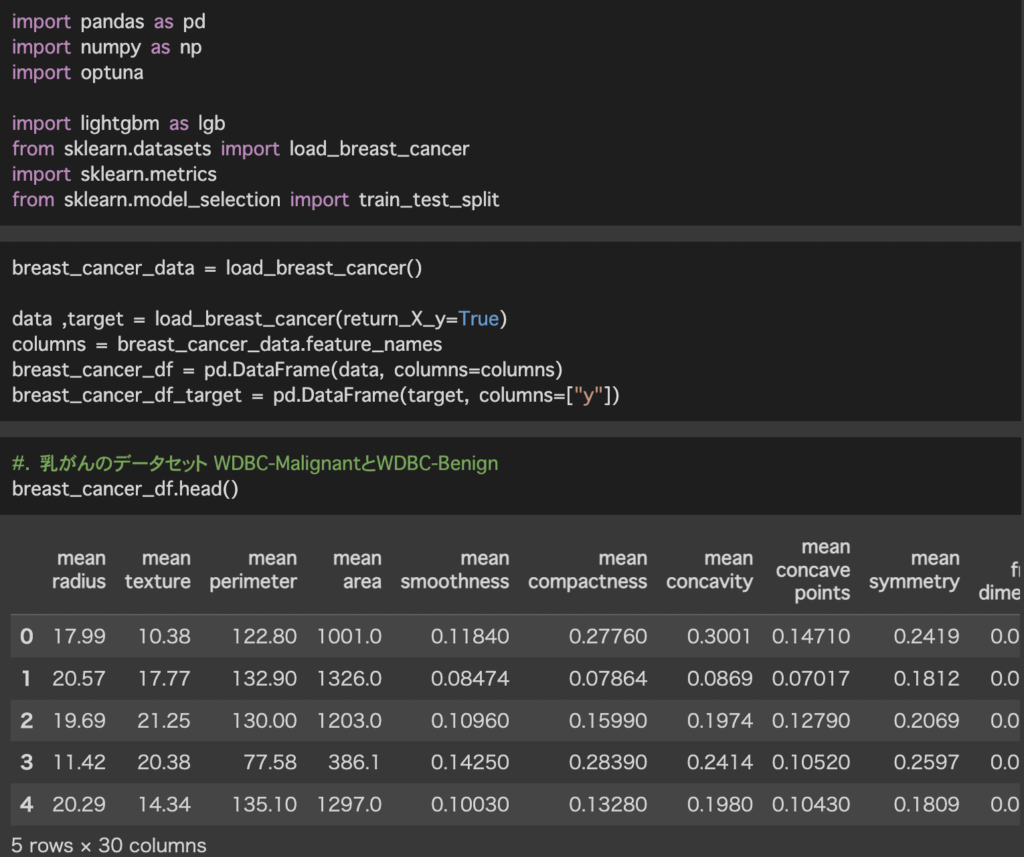
sklearnのdatasetsにあるload_breast_cancerを用います。
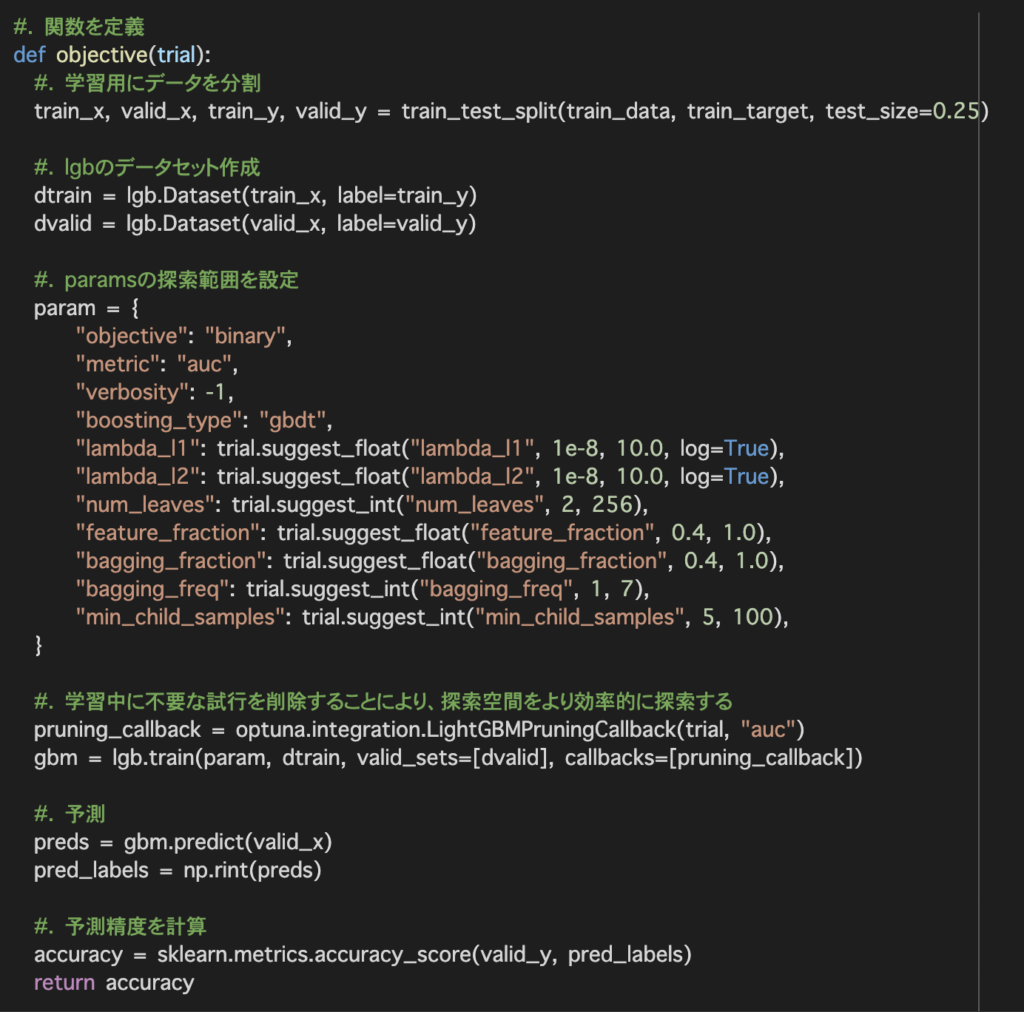
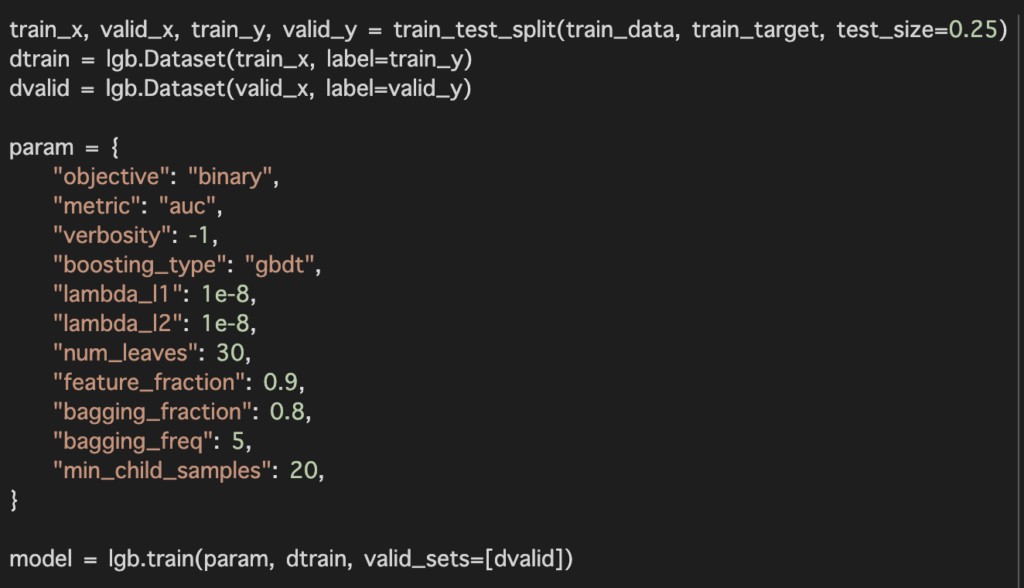
paramでハイパラメータの探索範囲を設定しています。
次にoptuna.integration.LightGBMPruningCallbackを用いて不要な試行を削除することにより探索空間をより効率的に探索を行います。
最後に学習したモデルを用いて検証データの予測を行い予測精度を計算します。

study.optimizeは試行回数を設定しobjective関数を最適化し、n_trials引数に試行回数を実行します。
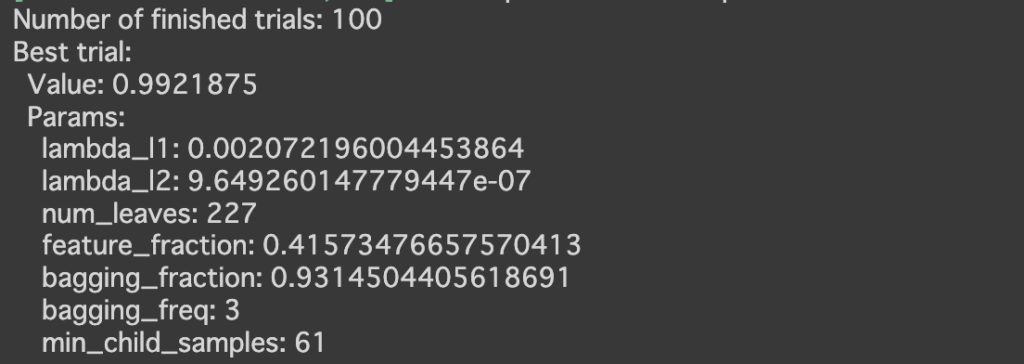
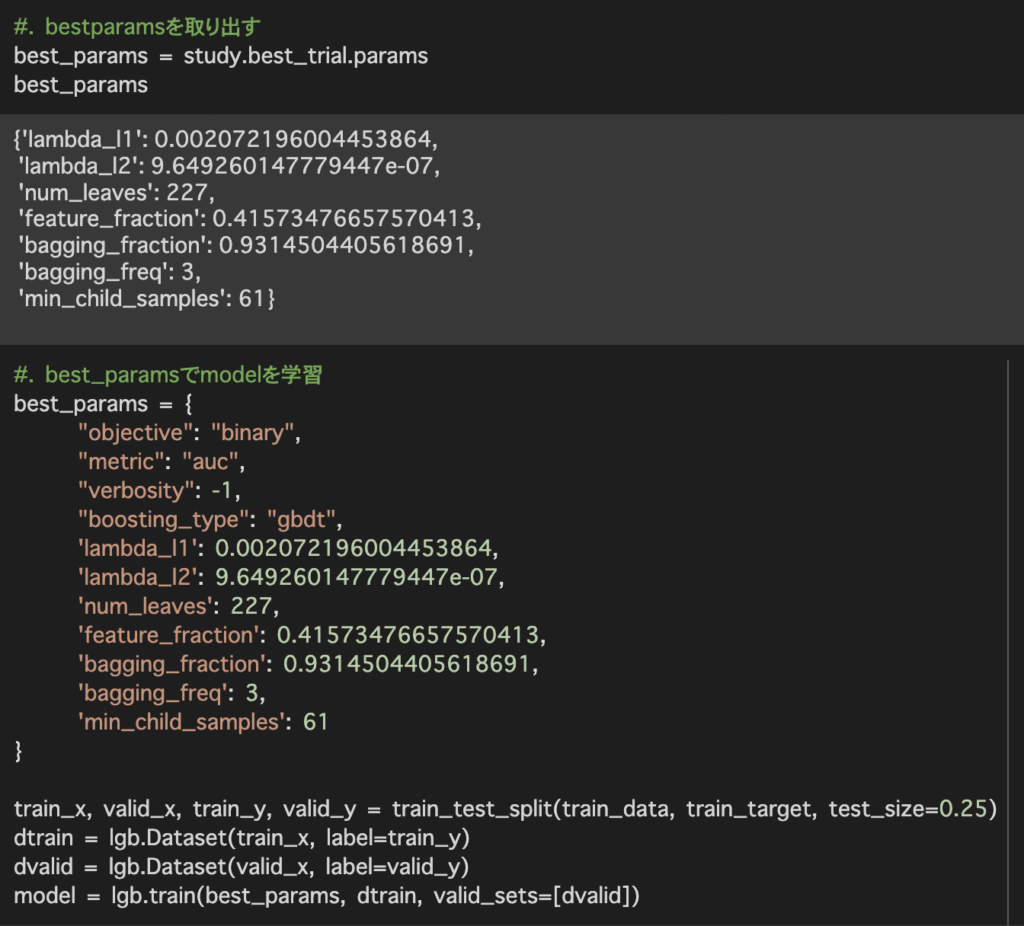
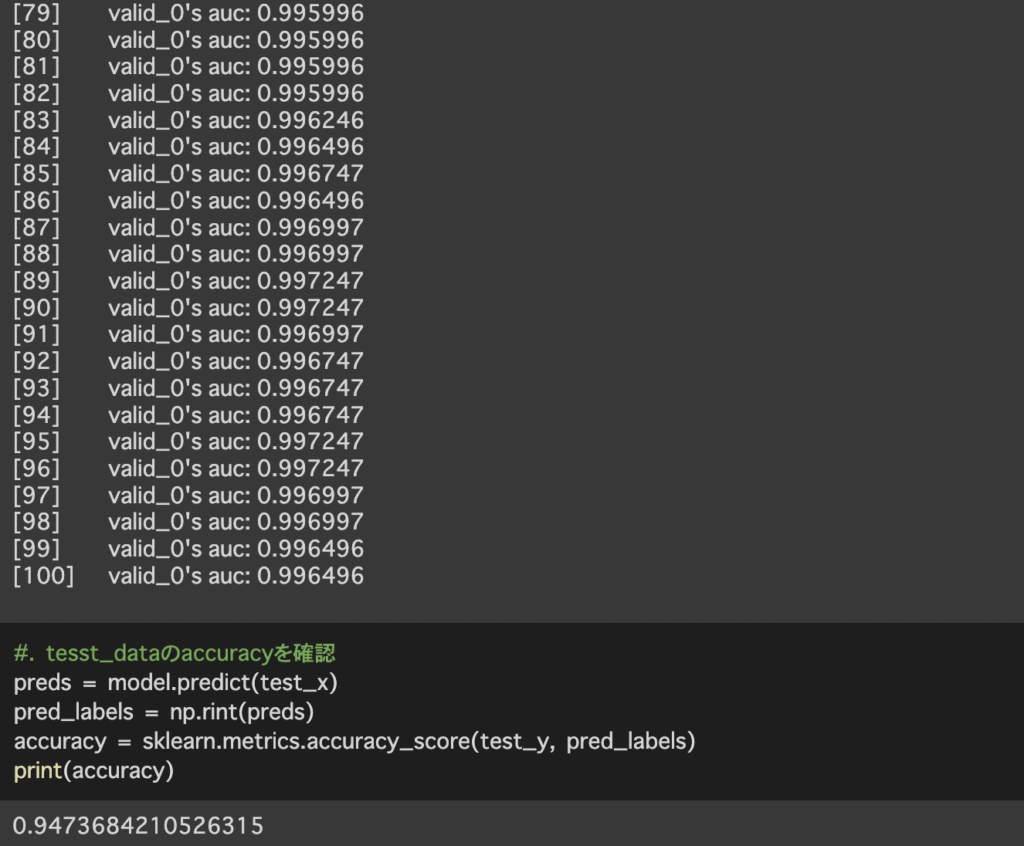
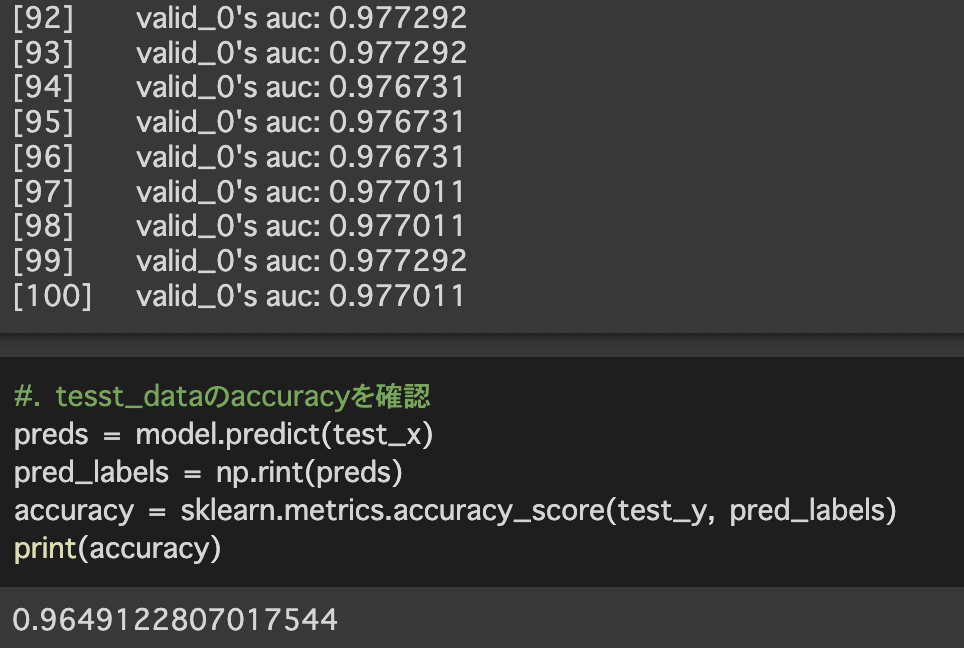
今回用いたモデルは大規模データに有効なlightgbmを使用しています。そのためデータ数が少ない今回のデータセットでは十分なパフォーマンスを発揮できなかった可能性が考えられます。
データセットや使用するモデルによって必ずしも最適な組み合わせが得られるわけではないと考えられる。そのため、どのような場合に用いるかは検討する必要があると考えられます。
○最後に
このような形で分析した結果や試してみたことを週に1回(目標)ペースで掲載しています。データ分析のキャリアを歩み始めたのですが、データの解釈、分析力が低いと感じ今回、このような形でアウトプットをしていくことにしたため、ぜひ、アドバイスやご指摘をいただけると幸いです。
コメントを残す